多次元尺度法をやってみた#1
多変量解析を行う際に、主成分分析は多変数データの情報を要約し、その本質を捉えるためのツールとなる。分散が最大となる方向を見つけ出し、それを新たな軸としてデータを変換することによって、データの次元を減らすことが可能となり、データの解釈が容易となる手法だ。一方、生物群集の多変量解析では、類似度を直接利用してデータ配置を行いたい、主成分分析で用いられる線形性の仮定が生物群集の分析にそぐわないといった理由から多次元尺度法(MDS:multi-dimensional scaling)が用いられることが多い(加藤,1996)らしい。
というわけで、カワバタモロコの採捕DATAからMDSをやってみた。MDSの計算方法はいくつか知られている(土井・岡村,2011)らしく、詳細は割愛したい。Chao指数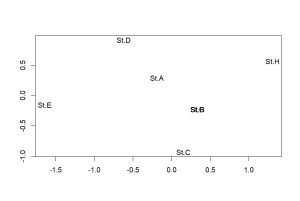 によるNMDSをやってみると、図の通りとなった。種構成と数量から見るカワバタモロコ群集の空間配置はSt.BとSt.FとSt.Gが似通っていることになった。これらの生息池の共通点はカワバタモロコしかいないこと。解析の妥当性が分かる。ということは、観測変数を多くしてMDSを利用することで、カワバタモロコが増える池と増えない池の特性が見えてきそうだ。
によるNMDSをやってみると、図の通りとなった。種構成と数量から見るカワバタモロコ群集の空間配置はSt.BとSt.FとSt.Gが似通っていることになった。これらの生息池の共通点はカワバタモロコしかいないこと。解析の妥当性が分かる。ということは、観測変数を多くしてMDSを利用することで、カワバタモロコが増える池と増えない池の特性が見えてきそうだ。
※土井秀幸・岡村寛.2011.生物群集解析のための類似度とその応用:Rを使った類似度の算出、グラフ化、検定.日本生態学会.61:3-20
※加藤和弘.1996.生物群集の多変量解析とその地域環境計画への応用.ランドスケープ研究.60:46-55
※Rコード
rm(list=ls())
library(MASS)
library(vegan)
data<-read.csv(“table.csv”,row.names=1)
head(data)
chmds<-metaMDS(data,dist=”chao”,k=2,zerodist=”add”)
plot(chmds$points,type=”n”,xlab=””,ylab=””)
text(chmds$points,dimnames(data)[[1]])

