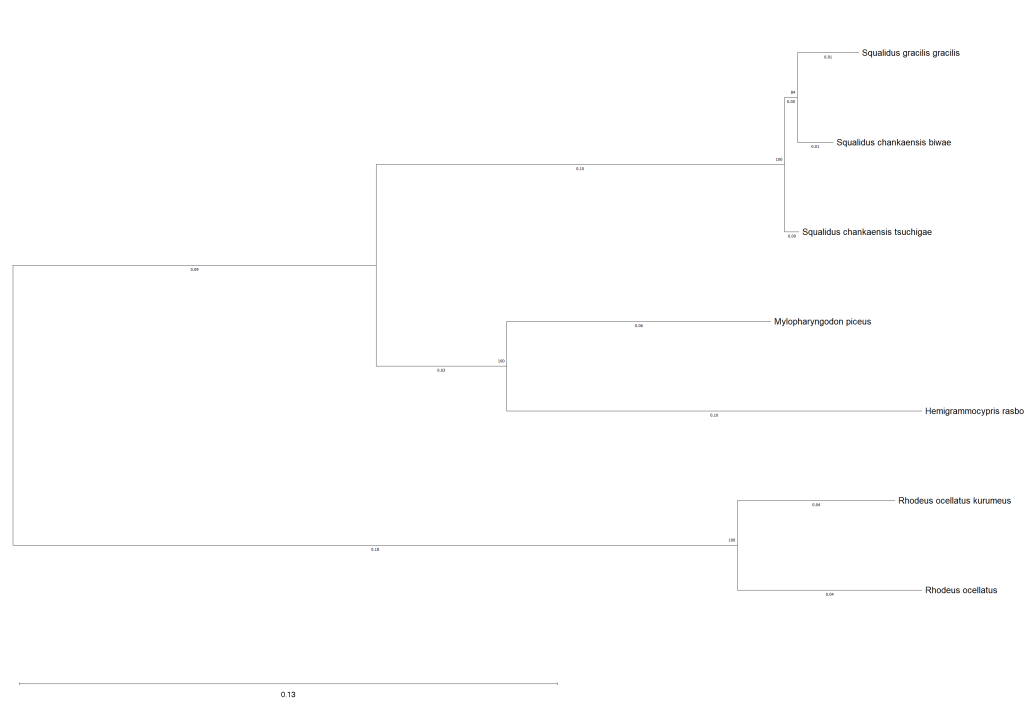
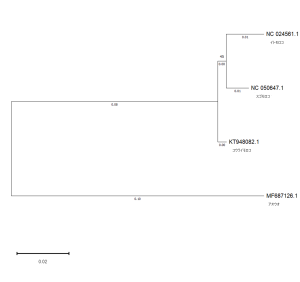
系統樹を描いてみた#2
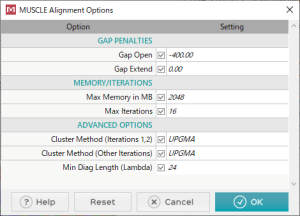 昨日に引き続き、系統樹を描いてみた。今日はニッポンバラタナゴ、タイリクバラタナゴ、カワバタモロコを加える。計算時間も適度に長くなる。Align DNAの設定は左図の通り。クラスタリングの方法は、UPGMA(unweighted pair group method with arithmetic mean)。平均距離法と呼ばれ、段階的探索法の一種で有根系統樹を作成する方法だ。各配列の距離行列を元に最近縁の配列を結合してクラスターを形成し、そのクラスターと他の配列との距離はクラスター内の各配列と対象の配列との距離の平均値とする。次に、距離の近い配列(またはクラスター)同士の結合を繰り返すことで、一つの有根系統樹を作成する。計算は容易であるが、進化速度が一定であると仮定するために系統間で進化速度が異なる場合は誤推定を行ってしまう欠点をもつらしい。
昨日に引き続き、系統樹を描いてみた。今日はニッポンバラタナゴ、タイリクバラタナゴ、カワバタモロコを加える。計算時間も適度に長くなる。Align DNAの設定は左図の通り。クラスタリングの方法は、UPGMA(unweighted pair group method with arithmetic mean)。平均距離法と呼ばれ、段階的探索法の一種で有根系統樹を作成する方法だ。各配列の距離行列を元に最近縁の配列を結合してクラスターを形成し、そのクラスターと他の配列との距離はクラスター内の各配列と対象の配列との距離の平均値とする。次に、距離の近い配列(またはクラスター)同士の結合を繰り返すことで、一つの有根系統樹を作成する。計算は容易であるが、進化速度が一定であると仮定するために系統間で進化速度が異なる場合は誤推定を行ってしまう欠点をもつらしい。
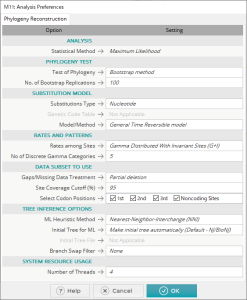 PHYLOGENY(系統発生)の設定は左図のようにした。Maximum Likelihood(最尤推定)が一般的とのことなので、そのように設定する。最尤推定は、母数の点推定をする一つの方法だが、これは母集団の分布の形がわかっているがその母数が未知であるときに、標本値からその母数を決めようとする方法。生物学では塩基やアミノ酸配列のような分子データの置換に関する確率モデルに基づいて系統樹を作成する際に、一番もっともらしくデータを説明する樹形を選択するための有力な方法として利用される。Bootstrap検定は、1000回くらいはしたほうが無難なようだが、100回でも構わないらしいので、今回はそうする。
PHYLOGENY(系統発生)の設定は左図のようにした。Maximum Likelihood(最尤推定)が一般的とのことなので、そのように設定する。最尤推定は、母数の点推定をする一つの方法だが、これは母集団の分布の形がわかっているがその母数が未知であるときに、標本値からその母数を決めようとする方法。生物学では塩基やアミノ酸配列のような分子データの置換に関する確率モデルに基づいて系統樹を作成する際に、一番もっともらしくデータを説明する樹形を選択するための有力な方法として利用される。Bootstrap検定は、1000回くらいはしたほうが無難なようだが、100回でも構わないらしいので、今回はそうする。